朝っぱらから正気を疑うようなことが起きた。statsmodelsは0.13.0より前のバージョンだと、BICの計算式が
BIC = deviance – df_resid * log(nobs)
になっている。僕がいつまでも古いバージョンを使っているのも悪いんだけど。
公式のReferencesにも

という文言が入っている。
ちなみに僕が現状で使っているのは、
import statsmodels
print(statsmodels.__version__)

もちろんアップデートすればいい話なんだけど、論文を書いている途中でパッケージを0.12.x→0.13.5にアップデートするのは結構な危険が伴う。値が変わるのがBICだけとは限らない。
というか、まあ実はGLMのresultはそのままそれに対応するためにBICを2種類呼べるようになっている。たとえばGLMの結果を
import statsmodels.api as sms
model = sms.GLM(Y, X, families=sms.families.Gamma(link=sms.genmod.families.links.log()))
res = model.fit()
みたいな感じでstatsmodels.genmod.generalized_linear_model.GLMResultsクラスが返ってきたら、通常AICやBICを表示させるときには
print(res.summary2())
で表示させるだろうし、その中から個別の値(たとえばパラメータ推定値とか)なんかを引っ張ってくるときには
res.params
res.aic, res.bic
あたりで取り出すわけなんだけど、実はbicはres.bic以外にもres.bic_devianceとres.bic_llfでも呼べるようになっていて、このbic_llfがlog-likehoodベースで計算したBICに該当する。その定義については上と同じページ内に、
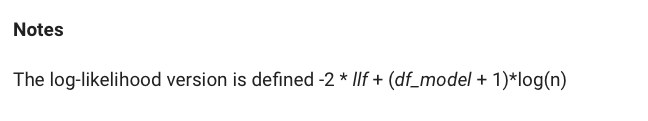
で計算されていることが示されている。
まあ対数尤度は既に計算されているわけだし、ここまでくるとAIC, BICの算出に関してはサンプルサイズと自由度から自分で出したほうがいいまである。
パッケージに頼ると本当にこういうことが起きる。特にPythonは工学系分野の色が強くてところどころ僕らの常識は共有されていない感じがする(それはstatsmodelsにTobitが存在しないことにも言える)。大人しくSASを使うべきなんだろうなあ。Googleに追加料金を払ったらColab上でSASを利用できるように!みたいなこと起きないのかな。絶対起きないだろうな。
みなさまもお気をつけあれ。

