基本的には全部Pythonでスクラッチから実装するので(高精度で計算がしたければSASもMATLABもあるし)Rを使うことって本当にたまにしかないんですよね。しかし、たまにしか使わないからこそ、そのたまのタイミングでエラーばっか吐いて一向に動かないということは往々にして起きうるのであった。
Colabのカーネルとしては
!jupyter kernelspec list
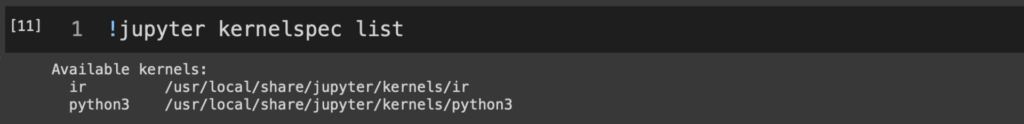
ご覧の通り既にRとPythonが用意されていて(昔はSwiftも入っていたらしいが)、使おうと思えばすぐ使える。
本当にちょろっと触るだけなら%%scriptコマンドでもできる。
%%script R --vanilla --quiet
1 + 1
とかでできるんだけど、これはバッチを走らせるようなもんで、要はスクリプトを走らせ終わったらRの処理は完全に終わるので、Jupyterのインタラクティブさは全くもって失われる。
インタラクティブさを維持しつつRとPythonの環境を共存させる一番簡単な方法はrpy2なので、とりあえずこいつのextentionを有効にする。
%load_ext rpy2.ipython
これの利点は、冒頭に%%Rコマンドが入っているセルのみ自動的にRのコードとして処理されること。爆発的に便利。
ということで久々に使おうとしたら、
%%R
library(data.table)
NotImplementedError: Conversion ‘py2rpy’ not defined for objects of type ‘<class ‘str’>’
このタイミングでこのエラーが出るのはおかしい。確かにpythonからrpyには文字型をはじめとして”変”なObjectを渡そうとするとこの類のエラーが出る。ただ、今回の場合はまた何の変数も渡していない。
調べたところ、直近1ヶ月前にStackOverflowに質問と良回答が上がっていた。
This is a problem with newer versions. You must downgrade rpy: run in a cell
つまるところバージョンの関係だった。ということでrpyを有効化する前にpipで指定バージョンを読み出すように書き換える。とりあえずランタイムを再起動してから、
!pip install rpy2==3.5.1
%load_ext rpy2.ipython
これでPython側のオブジェクトも自由に渡せるR環境ができる。
import pandas as pd
dat = pd.DataFrame({
'a':[1,2,3],
'b':[4,5,6],
}, index=[0,1,2])
%%R -i dat
dat
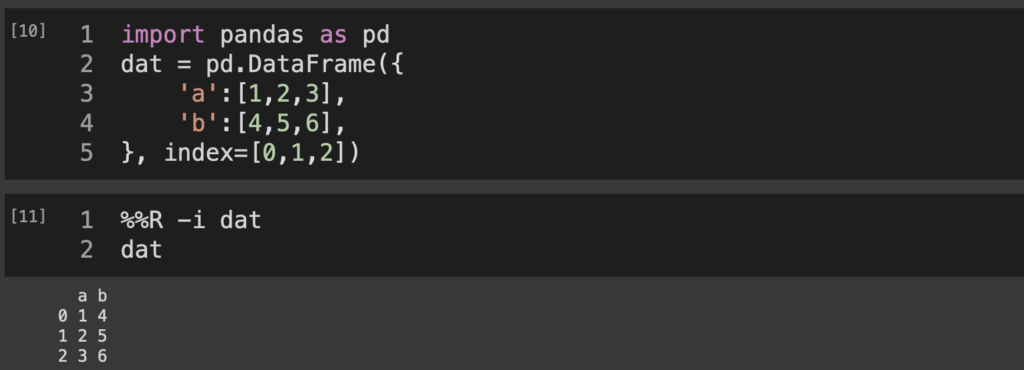
できた。これぐらい環境を行き来できると本当にいいですね。

